
Antibióticos actuales
Antibióticos clásicos
Podemos ver una rápida clasificación de los antibióticos según donde actúan en la bacteria.
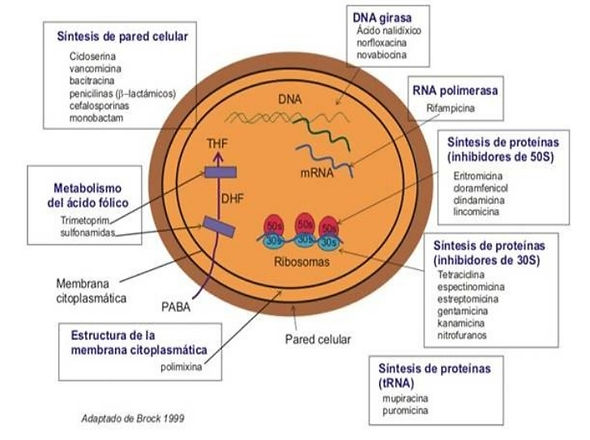
Imagen adaptada del Brok (1999)
Los que actúan sobre la síntesis de la pared celular son los antibióticos más conocidos, y entre ellos esta el primero que se descubrió (por serendipia), la penicilina. Aunque las bacterias han ingeniado mil y un mecanismos para evadirse de ellos y evitar la destrucción de su preciada pared celular.
Por otro lado, tenemos un grupo que interesa evitar la generación de resistencias por parte de las bacterias dado que son muy efectivos. Este grupo son las quinolonas, más concretamente las fluoroquinolonas (como la norfloxacina, novabiocina, etc.) Actúan en la DNA girasa y son eficaces contra bacterias Gram positivas y Gram negativas, como también en micobacterias, micoplasma, clamidias, legionelas y brucella. Por lo que es un antibiótico muy preciado que se usa en casos muy concretos. [4]
Resistencias:
También es interesante conocer los mecanismos generales de generación de resistencia a los antibióticos de las bacterias. Conociendo a tu enemigo, se puede combatir mejor contra él, pudiendo así bloquear las rutas alternativas que realiza la bacteria para contrarestar el antibiótico. Como sería el caso de el ácido clavulánico, que ayuda a la no inhibición de la penicilna, haciendo muy interesante la combinación de estos dos fármacos, lo que nos lleva a pensar que investigar en combinaciones de fármacos puede conllevar a unos efectos positivos.
Los principales mecanismos de resistencia son:
Producción de enzimas inactivadoras:
Alteración del sitio de acción o de fijación:
Las β - lactamasas para penicilinas y cefalosporinas.
La acetiltransferasa para el cloranfenicol.
La fosforilación o acetilación para los aminoglucósidos.
Cambios en la subunidad 30S para los aminoglu-cósidos.
Cambios en las subunidad 50S para la eritromicina.
Alterar ARN polimerasa dependiente de ADN para la rifampicina.
Expulsión del antibiótico para las tetraciclinas.
Disminución de la captación para la ampicilina.
Cambios de la envoltura para aminoglucósidos y β - lactámicos.
Disminución de la acumulación del fármaco en la bacteria:
Síntesis de enzimas insensibles, como la dihi- dropteroato que tiene una baja afinidad para las sulfamidas. [5]
Plásmidos
Desarrollo de vías alternativas:
Plásmidos
Plásmidos
Codificado por:
Plásmidos o trasposones
Mutación genética
Plásmidos
Mutación genética
Plásmidos
Mutación genética
Mutación genética
Según la Organización Mundial de la Salud (OMS), más del 95% de cepas de Staphylococcus aureus en todo el mundo son ahora resistentes a la penicilina, y hasta el 60% son resistentes a su derivado, la meticilina. La resistencia se está extendiendo también por patógenos como los neumococos y la tuberculosis. Es alarmante que en Japón, Europa y los Estados Unidos, ha habido al menos cuatro casos de S. aureus resistentes a vancomicina, el antibiótico de último recurso en el tratamiento de las infecciones que amenazan con la vida [6]. Este creciente problema de salud pública estimula los esfuerzos para descubrir nuevos agentes antibacterianos con mecanismos radicalmente diferente de los compuestos existentes. Varias empresas pequeñas están probando nuevas clases de agentes antibacterianos que se dirigen a las membranas celulares bacterianas, a los mecanismos de adherencia o en la expresión génica.
Y es que casi no hay nuevas moléculas desde hace más de 30 años. Aunque entre ellas podemos encontrar:
Antibióticos de nueva generación

Tabla resumen sobre los nuevos antibióticos. [7][8][9][10]
Investigaciones recientes y aplicaciones
Aparte de los antibióticos de nueva generación expuestos, se comentarán dos investigaciones recientes; el descubrimiento de una nueva molécula (Teixobactin), actúa como un antibiótico del cual no se ha reportado aún resistencias y ha sido descubierto de una forma muy interesante, como también de las primeras estructuras cristalinas de las proteínas unidas covalentemente a penicilina inactivadas.
El grupo de Losee L. Ling y Tanja Schneider han publicado este enero del 2015 en el Nature un artículo donde muestran un nuevo antibiótico (TEIXOBACTIN) [11]. Teixobactin actúa inhibiendo la síntesis de la pared celular de las bacterias mediante la unión a un motivo altamente conservado de lípido II (precursor de peptidoglicano) y lípido III (precursor del ácido teicoico de la pared celular). Este grupo no ha obtenido ningún mutante de Staphylococcus aureus ni de tuberculosis micobacteriana resistente a Teixobactin. Por lo que las propiedades de este compuesto sugieren un camino hacia el desarrollo de antibióticos que son propensos a evitar el desarrollo de resistencias. Una cuestión muy interesante para poder paliar y erradicar las infecciones bacteriana.
Los autores vieron que Teixobactin tenía una actividad excelente contra bacterias Gram positivas, incluso con las cepas resistentes a otros antibióticos:
Ellos descubrieron esta molécula a través del isolation chip (Ichip). Un dispositivo que permite cultivar unas 10.000 bacterias en su entorno natural, en este caso, el suelo. Una vez realizado el cultivo, los extractos de estas células fueron expuestos a Staphylococcus aureus, para poder así identificar los compuestos con posibilidad bactericida contra este patógeno. De esta manera que se identificaron varias bacterias del suelo que generaban moléculas que funcionan como antibiótico. Al final los autores identificaron la bacteria Elephtheria terrae como productor del nuevo antibiótico, la Teixobactin.

Teixobactin
IChip, cultivando las bacterias en su medio natural, el suelo. [12]
IChip
Ciertamente el Ichip es un dispositivo interesantísimo para el descubrimiento de nuevos antibióticos producidos por bacterias. El hecho de que se cultiven en su ambiente nos abre un gran abanico de posibilidades y nos lleva a pensar que se podrían cultivar bacterias de lo más inhóspitas capaces de producir infinidad de moléculas efectivas como antibióticos.

Funcionamiento y pasos del Ichip hasta que se logró hayar la Teixobactin. [13]
En 2011, el grupo de Seungil Han consiguió las primeras estructuras cristalinas de la proteína unida covalentemente a penicilina (PBP) de Acinetobacter baumannii, inactivadas por cuatro antibióticos β-lactámicos [14]. Estos datos estructurales, biofísicos y computacionales pueden ayudar en un futuro diseño de agentes antimicrobianos.
Cristalización de PBP
Se puede ver en la imagen [14] las estructuras globales de Acinetobacter PBP1a y PBP3. Donde (A) es la estructura de AbPBP1a (Penicillin-Binding Protein 1a de Acinetobacter baumannii) y (B) la estructura de AbPBP3 (Penicillin-Binding Protein 3 de Acinetobacter baumannii) con el dominio N-terminal en rosa y el dominio C-terminal en azul. También vienen determinados los dominios, donde TG es el dominio transglicosilasa, OB el dominio de unión de oligonucleotidos/oligosacaridos, TP el dominio transpeptidasa y nPB el dominio de no unión a la penicilina.

Realizar cristalización de proteínas es de vital importancia para comprender el funcionamiento de estas e incluso diseñar métodos para inhibir o desestructurar proteínas bacterianas. O por el contrario, conocer más sobre proteínas de nuestro propio organismo y concebir así novedosos antimicrobianos. Ya se están llevando a cabo cristalizaciones de las RNasas humanas, unos péptidos antimicrobianos muy convenientes.
Referencias:
^ 4. J.A. Linder, E.S. Huang, M.A. Steinman, R. Gonzales, R.S. Stafford. Fluoroquinolone prescribing in the United States: 1995 to 2002. The American Journal of Medicine (2005); 118,3:259–68
^ 5. Katzung, Bertram G. Basic & Clinical Pharmacology (2007, 9 edición). McGraw-Hill.
^ 6. H. Breithaupt. The new antibiotics. Nature biotechnology (1999);1165-1169
^ 7. J. Pogliano, N. Pogliano, J.A. Silverman. Daptomycin-Mediated Reorganization of Membrane Architecture Causes Mislocalization of Essential Cell Division Proteins. Journal of Bacteriology (2012) 194,17: 4494–4504
^ 8. K.O. Cleveland and M. S. Gelfand. Daptomycin-Nonsusceptible Enterococcal Infections; Infect Dis Clin Pract (2013); 21:79-84.
^ 9. V. Kothary, E. J. Scherl, B. Bosworth, Z. Jiang, H.L. DuPont, J. Harel, K.W. Simpson and B. Dogan. Rifaximin Resistance in Escherichia coliAssociated with Inflammatory Bowel Disease Correlates with Prior Rifaximin Use, Mutations in rpoB, and Activity of Phe-Arg-β-Naphthylamide-Inhibitable Efflux Pumps. Antimicrob. Agents Chemother (2013); 57:2811-817
^ 10. N. Scheinfeld. Telithromycin: A brief review of a new ketolide antibiotic. J Drug Dermat (2004); 3:409-13
^ 11. L. L. Ling, T. Schneider, A. J. Peoples, A. L. Spoering, I. Engels, B. P. Conlon, A. Mueller, T. F. Schäberle, D. E. Hughes, S. Epstein, M. Jones, L. Lazarides, V. A. Steadman, D. R. Cohen, C. R. Felix, K. A. Fetterman, W. P. Millett, A. G. Nitti, A. M. Zullo, C. Chen and K. Lewis. A new antibiotic kills pathogens without detectable resistance. Nature (2015); 517:455–459.
^ 12. Portal de noticias del nature. Información sobre el Ichip. Visitado en abril 2015: URL: http://www.nature.com/news/promising-antibiotic-discovered-in-microbial-dark-matter-1.16675
^ 13. G. Wright. Antibiotics: An irresistible newcomer. Nature (2015); 517:442-444
^ 14. S. Han, N. Caspers, R. P. Zaniewski, B. M. Lacey, A. P. Tomaras, X. Feng, K. F. Geoghegan and V. Shanmugasundaram. Distinctive Attributes of β-Lactam Target Proteins in Acinetobacter baumannii Relevant to Development of New Antibiotics. J.Am. Chem. Soc. (2011); 133:20536–20545

